최근에 바이오인포 쪽 랩에서 학부 연구생을 하게 되었는데, 생각보다 한국어 자료가 많지 않아서 남겨보는 기록!
틀린 설명이 있을 수도 있음 주의
DEG 분석이란?
DEG는 Differentially Expressed Gene의 약자!
유전자의 발현값을 측정하고 통계적 과정을 거쳐 대조군과 비교군 간에 발현이 유의한 유전자 후보군을 선정하는 방식이다. (출처)
DEG 분석을 위해서는 RNA-seq 데이터가 필요하다. DEG 분석을 위한 RNA-seq 데이터를 만드는 방법은 여러 가지가 있겠지만 내가 사용한 방법은 다음과 같다.
1) RNA-seq를 통해 얻은 fq 파일에서 필요 없는 adapter 등의 서열을 잘라냄.
2) TopHat2를 이용한 mapping 진행.
3) Cufflinks, Cuffdiff 등의 프로그램을 이용하여 발현량을 계산.
4) 데이터를 파싱 및 가공 (-> 이 과정에서 이미 작성된 코드를 이용했음)
4번 과정까지 끝내면 우리는 replicate 별 유전자의 발현량 정보를 이용하여 유의확률이나 FPKM, Fold Change 등의 값을 찾을 수 있다.
(FPKM = Fragments Per Kilobase of transcript per Million - transcript당 fragment의 수)
(Fold Change = Transgenic / Wild-type으로 계산)
Volcano Plot
Volcano Plot은 scatter plot의 일종이라고 생각하면 된다. 시키는 대로 그리면 화산이 분출하는 듯한(!) 모양이어서 Volcano Plot이다.
x축은 log2(Fold Change) 값을, y축은 -log(p-value)값을 나타낸다.
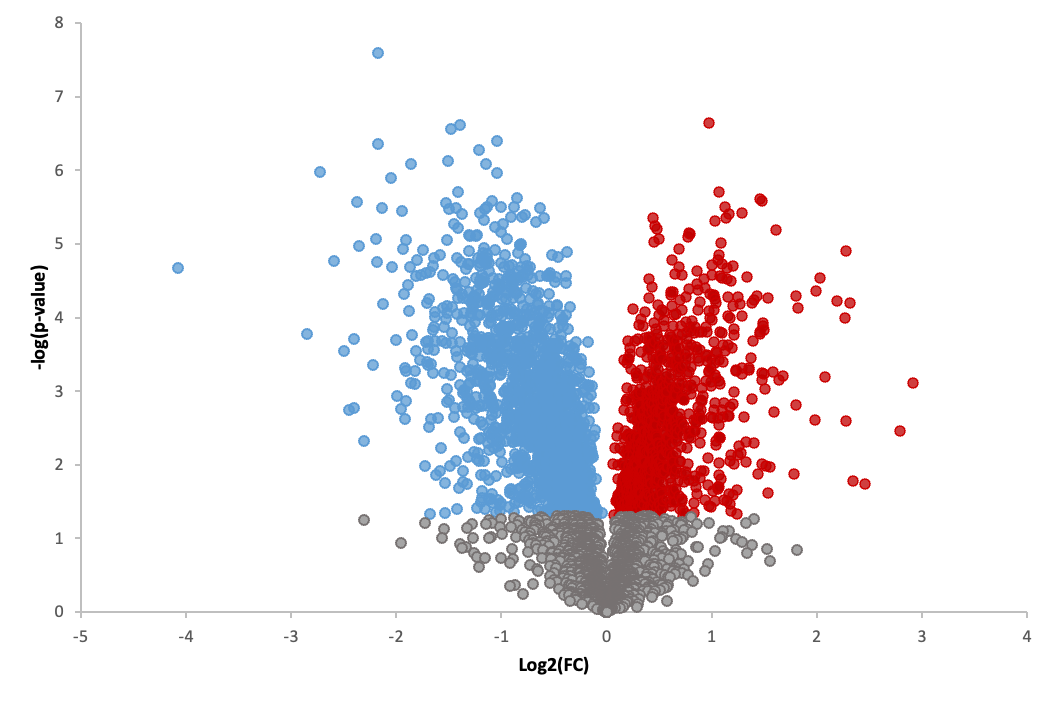
대충 그리면 이런 느낌이다. x=0을 기준으로 왼쪽은 log2(Fold Change) 값이 0보다 작으므로 down regulated gene, 오른쪽은 up regulated gene이라고 볼 수 있을 것이다.
보통 p<0.05를 유의하다고 보기 때문에 그 값을 만족 못하는 경우(회색 점들) 발현이 유의하다고 보지 않는다.
MA plot
RNA-seq의 Intensity-dependent ratio를 밝히고 시각화 하는데 매우 강력한 통계적 방법이다. (출처)
복잡하게 적혀있지만, 이 친구도 역시 scatter plot의 일종이라고 보면 될 것 같다.
x축에는 해당 유전자의 log2(FPKM)값의 평균을 나타내며, y축에는 log2(Fold Change)를 나타낸다. 예시 그림은 다음과 같다.
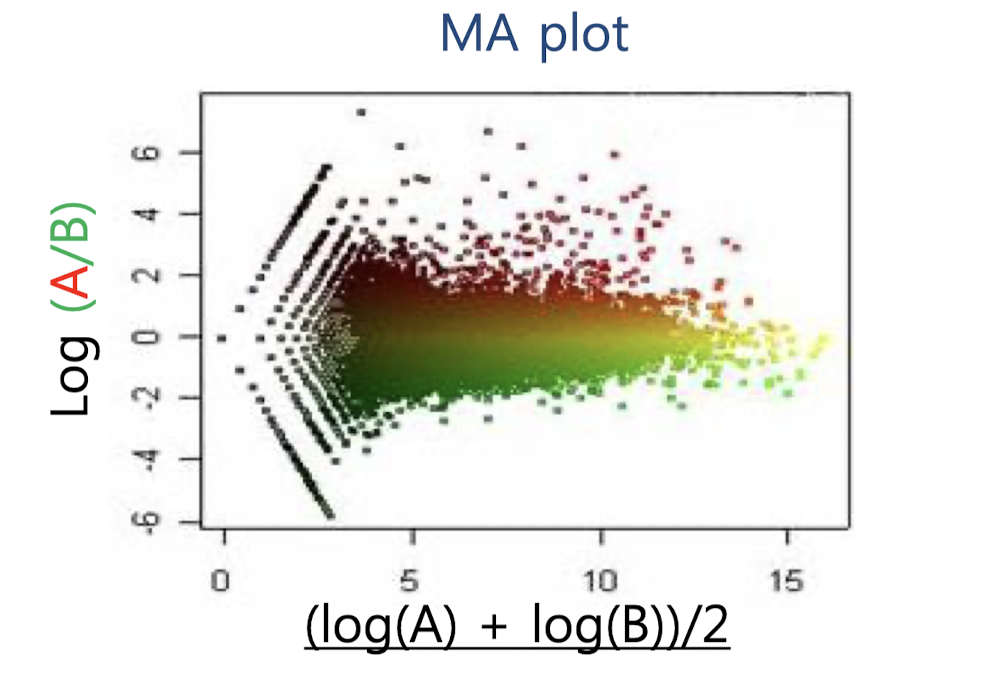
MA plot에서도 유의미(?)하지 않은 값들을 제거하는 과정을 거치는데 이 과정을 Volume Cut이라고 부른다고 한다.
무조건 들어맞는 기준은 없고 육안으로 살펴보면서 적당히 직선 x=n을 기준으로 오른쪽에 있는 값들만 사용할 수 있도록 해야한다.
보통은 MA plot을 먼저 그려서 volume cut을 정하고, 그 이후에 volcano plot을 그려서 Up/Down Regulated Gene을 구하는 듯...
그 다음엔 무엇을 해야 하지?
DEG 분석의 첫 걸음인 Up/Down Regulated Gene 찾기가 끝났다!
이제 이 정보를 DAVID, GOrilla, Revigo 등의 사이트에 입력해보면서 유전자 발현 차이를 확인해보면 된다.


